Lo studio Unipd sul meccanismo di interazione tra farmaci antivirali e la proteasi principale del SARS-CoV-2
01.12.2020
L’identificazione di un nuovo farmaco è un percorso lungo e complesso che richiede in media una decina di anni per essere portato a termine, necessita di ingenti risorse economiche e l’impiego di diverse competenze scientifiche trasversali. Una riduzione delle tempistiche, in particolar modo in uno scenario come quello attuale segnato dalla pandemia da Covid-19, porterebbe enormi vantaggi. I recenti sviluppi delle tecnologie informatiche unitamente ai progressi della chimica farmaceutica di frontiera hanno portato a “simulare l’intero processo di riconoscimento tra un farmaco e il suo bersaglio molecolare”. L’obiettivo è comprendere, appunto attraverso una accurata replicazione virtuale, cosa accade nell’organismo umano per poi tradurre questi risultati in azione terapeutica.
La sezione di Modellistica molecolare del Dipartimento di Scienze del farmaco dell’Università di Padova ha sviluppato una metodologia in grado, con tempi ragionevoli ed elevata accuratezza descrittiva, di seguire il percorso che porta un farmaco a incontrare e riconoscere quella particolare regione del bersaglio molecolare in cui il farmaco svolgerà la sua funzione.
Il lavoro del team guidato da Stefano Moro, professore del Dipartimento di Scienze del farmaco, sul meccanismo d’azione di un farmaco a livello molecolare è stato recentemente pubblicato sulla rivista «Scientific Reports» con il titolo con il titolo "Targeting the coronavirus SARS-CoV-2: computational insights into the mechanism of action of the protease inhibitors lopinavir, ritonavir and nelfinavir" (Giovanni Bolcato, Maicol Bissaro, Matteo Pavan, Mattia Sturlese, Stefano Moro).
Ad oggi non è disponibile alcun dettaglio molecolare riguardante il meccanismo d’azione di farmaci antivirali che possa supportare un loro impiego clinico nell’emergenza Covid-19. Storicamente la strategia sanitaria è stata quella di utilizzare precedenti evidenze cliniche efficaci in altre patologie e poi applicarle ai contesti nuovi di emergenza pandemica. Nella prima ondata di infezione da SARS-CoV-2 si è fronteggiato il virus con trattamenti farmacologici, già approvati per altre infezioni virali, da utilizzare in ambito emergenziale per la cura di Covid-19. In particolare, facendo tesoro delle evidenze della primissima pandemia da coronavirus (SARS, 2002-2003), i medici hanno adottato questo approccio terapeutico utilizzando farmaci antiretrovirali pensati per la cura dell’HIV raccogliendo indicazioni preliminari positive sull’efficacia del trattamento, restando comunque sempre in attesa di un vaccino efficace.
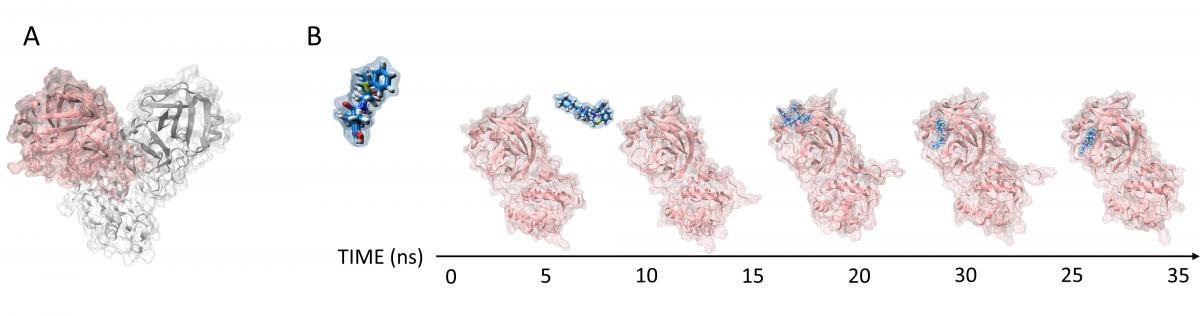
«La particolare tecnica che è stata sviluppata nel nostro laboratorio - dice Stefano Moro - si chiama dinamica molecolare supervisionata (SuMD) e ha trovato in questo periodo davvero complesso legato alla pandemia dovuta al coronavirus SARS-CoV-2 una sua applicazione nel descrivere a livello molecolare il riconoscimento tra una importante proteina del virus, la proteasi principale Mpro, e tre farmaci utilizzati in ambito clinico per il trattamento della patologia conseguente all’infezione virale. Di questi tre farmaci - continua Moro - mancava ancora una descrizione a livello molecolare del loro meccanismo di interazione con la proteasi Mpro».
In particolare questa tecnica consente di seguire come nel tempo il farmaco si avvicini alla sua proteina bersaglio, in quale regione della sua superficie avvenga il riconoscimento e quali siano le proprietà chimiche del farmaco che consentono a questo, dopo essere stato riconosciuto, di espletare la sua attività.
«L’applicazione di questa metodologia, oltre ad aver permesso di poter interpretare a livello atomico attraverso quale meccanismo avvenga il riconoscimento tra i tre farmaci (Lopinavir, Ritonavir e Nelfinavir) e la proteasi Mpro, getta le basi per una nuova fase di progettazione di farmaci candidabili per questa stessa proteina virale e per altre proteine che sono cruciali per il ciclo di replicazione di SARS-CoV-2 - affermano Giovanni Bolcato e Maicol Bissaro dottori di ricerca nella sezione di Modellistica molecolare del Dipartimento di Scienze del farmaco Unipd -. Con questa tecnica computazionale sarà possibile quindi suggerire nuove strutture chimiche che presentino una migliore efficacia terapeutica riducendo per quanto possibile gli effetti collaterali sull’organismo umano. Le principali ricadute dell’utilizzo di questa metodologia potranno supportare gli studi sperimentali nell’identificare precocemente quali tra tutti i possibili candidati farmaci presentino le caratteristiche ottimali per un riconoscimento efficiente del nostro bersaglio molecolare. Non solo, sarà possibile diminuire drasticamente - concludono Bolcato e Bissaro - il numero di composti chimici da introdurre nella filiera per lo sviluppo di un nuovo farmaco, riducendone di conseguenza tempi e costi associati. Infine si potranno avere indicazioni preliminari, ma accurate, del profilo di potenziale tossicità di questi farmaci applicando la stessa metodologia nei confronti di tutte quelle proteine che non dovrebbero contestualmente essere riconosciute dal medesimo composto e che devono quindi mantenere inalterata la loro funzionalità anche durante l’instaurarsi e l’evolversi della malattia».



